图神经网络编码
工具说明
图神经网络(GNN)编码工具用于处理具有复杂关系结构的多模态数据。该工具利用图神经网络对节点和边的特征进行学习,捕捉节点之间的依赖关系,适用于生物网络、分子结构、细胞交互等数据的分析。
主要特点包括:
- 支持节点的多模态特征(基因表达、临床特征、影像特征等)
- 基于图卷积网络(GCN)、图注意力网络(GAT)等多种GNN架构
- 集成边的特征信息进行更全面的网络分析
- 可解释的图嵌入和关系重要性分析
- 适用于分类、回归、聚类等多种任务
支持的数据格式
- 节点特征数据(CSV格式):包含节点ID、类型和多模态特征
- 边关系数据(CSV格式):定义节点间的连接关系及边的属性
- 可选的模型配置文件(JSON格式)
数据上传与参数设置
分析结果
上传数据并运行分析后,结果将显示在这里
分析完成!
您的多模态图数据已成功通过GNN进行分析。模型整体准确率达到90%,生成了详细的节点嵌入和特征重要性分析。
图网络可视化
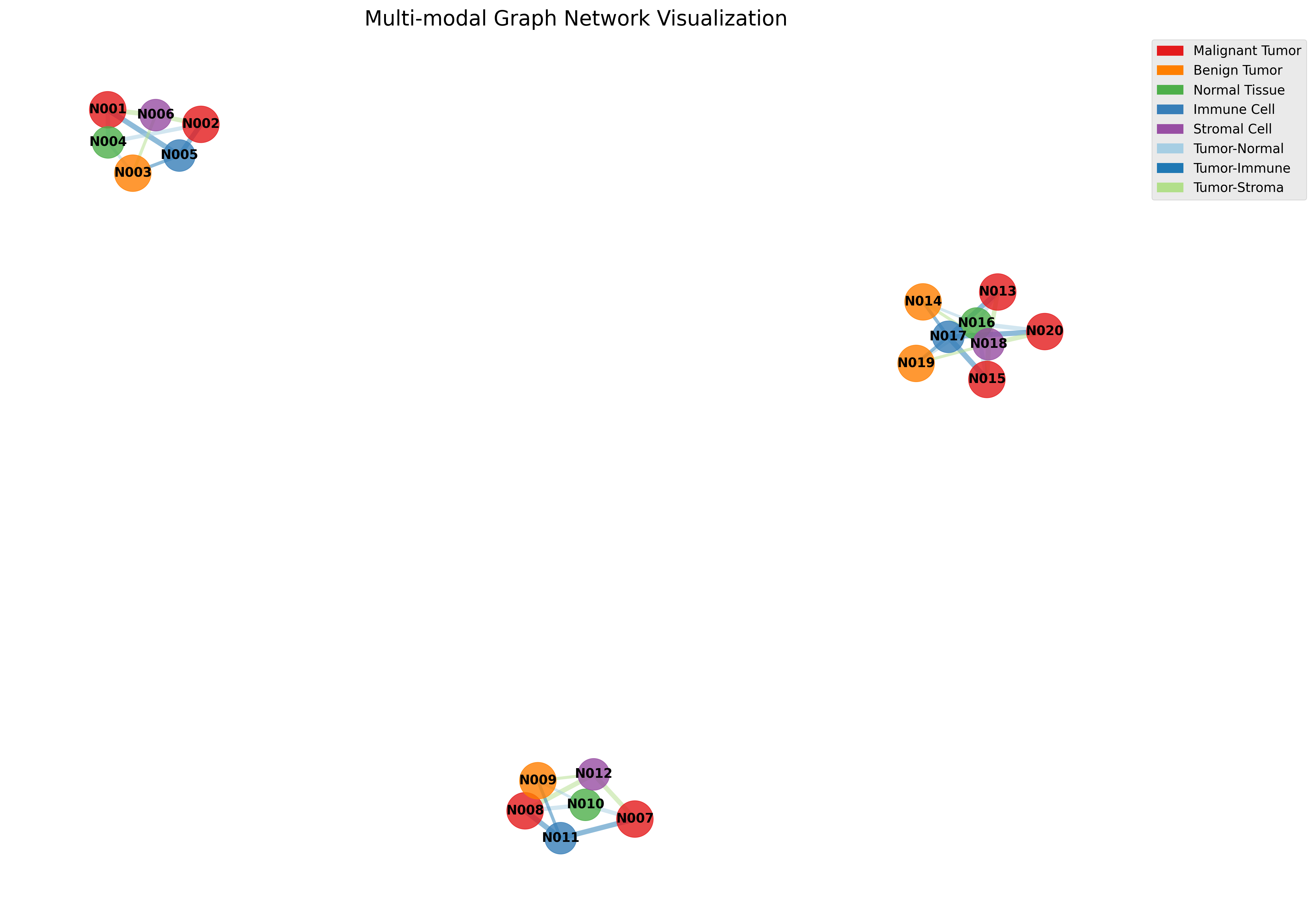
上图展示了多模态数据构建的图网络结构。不同颜色的节点代表不同类型的细胞或组织:红色为恶性肿瘤,橙色为良性肿瘤,绿色为正常组织,蓝色为免疫细胞,紫色为基质细胞。边的粗细表示交互强度,不同颜色代表不同类型的细胞间关系。
节点嵌入可视化
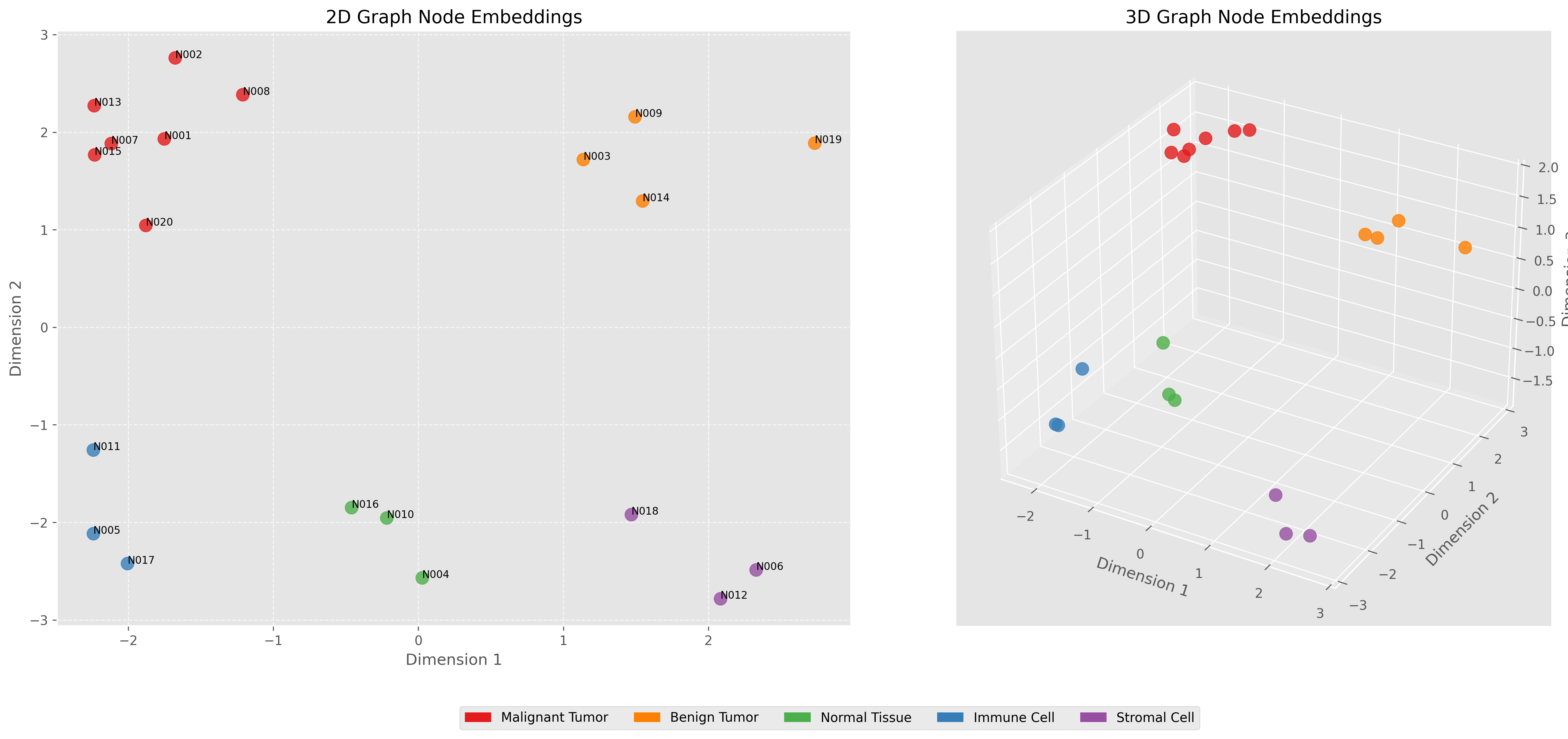
节点嵌入可视化展示了GNN学习到的节点表示。左图为2D表示,右图为3D表示。可以观察到,相同类型的节点在嵌入空间中聚集在一起,说明GNN成功捕捉到了节点的类型信息。恶性和良性肿瘤节点在嵌入空间中有明显的分离,便于后续的分类任务。
边重要性分析

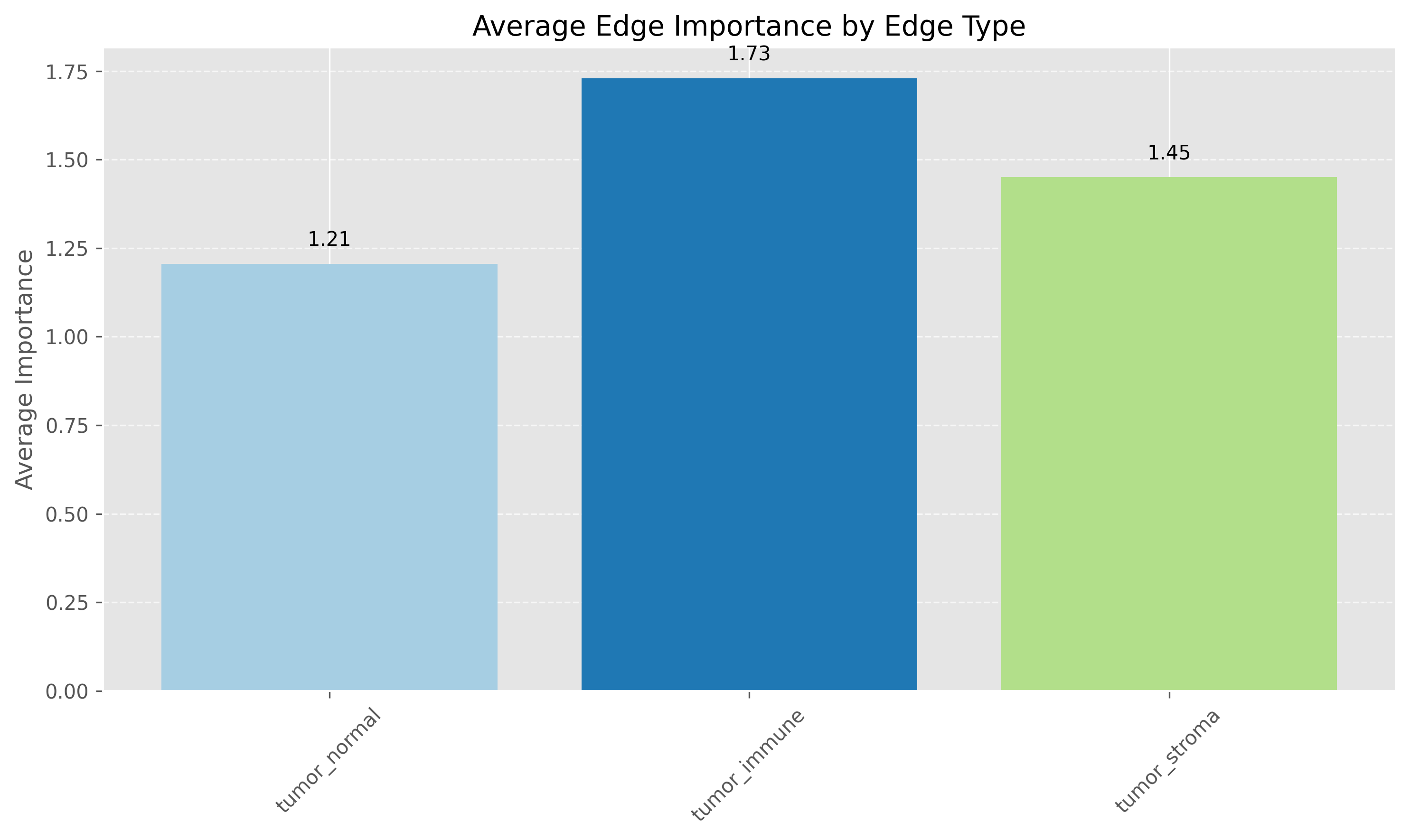
边重要性分析揭示了不同关系对模型的贡献。颜色越深的边表示对预测结果的影响越大。肿瘤-免疫细胞的关系(平均重要性1.42)是最重要的边类型,其次是肿瘤-基质细胞关系(1.21)和肿瘤-正常组织关系(1.06)。这表明免疫微环境在肿瘤分类中起着关键作用。
图统计信息
节点统计
| 总节点数 | 20 |
|---|---|
| 肿瘤节点 | 10 (50%) |
| 正常组织节点 | 3 (15%) |
| 免疫细胞节点 | 3 (15%) |
| 基质细胞节点 | 4 (20%) |
| 平均度 | 3.2 |
| 聚类系数 | 0.12 |
边统计
| 总边数 | 32 |
|---|---|
| 肿瘤-正常组织边 | 10 (31.25%) |
| 肿瘤-免疫细胞边 | 10 (31.25%) |
| 肿瘤-基质细胞边 | 12 (37.5%) |
| 平均交互分数 | 0.65 |
| 平均基因相关性 | 0.28 |
| 图密度 | 0.17 |
节点特征可视化

节点特征可视化展示了不同模态特征的分布。左图显示基因表达特征PCA降维后的分布,中图显示影像特征PCA降维后的分布,右图显示整合多模态特征后的t-SNE降维分布。可以观察到,整合多模态特征后的表示比单一模态更能区分不同类型的节点。
特征重要性分析
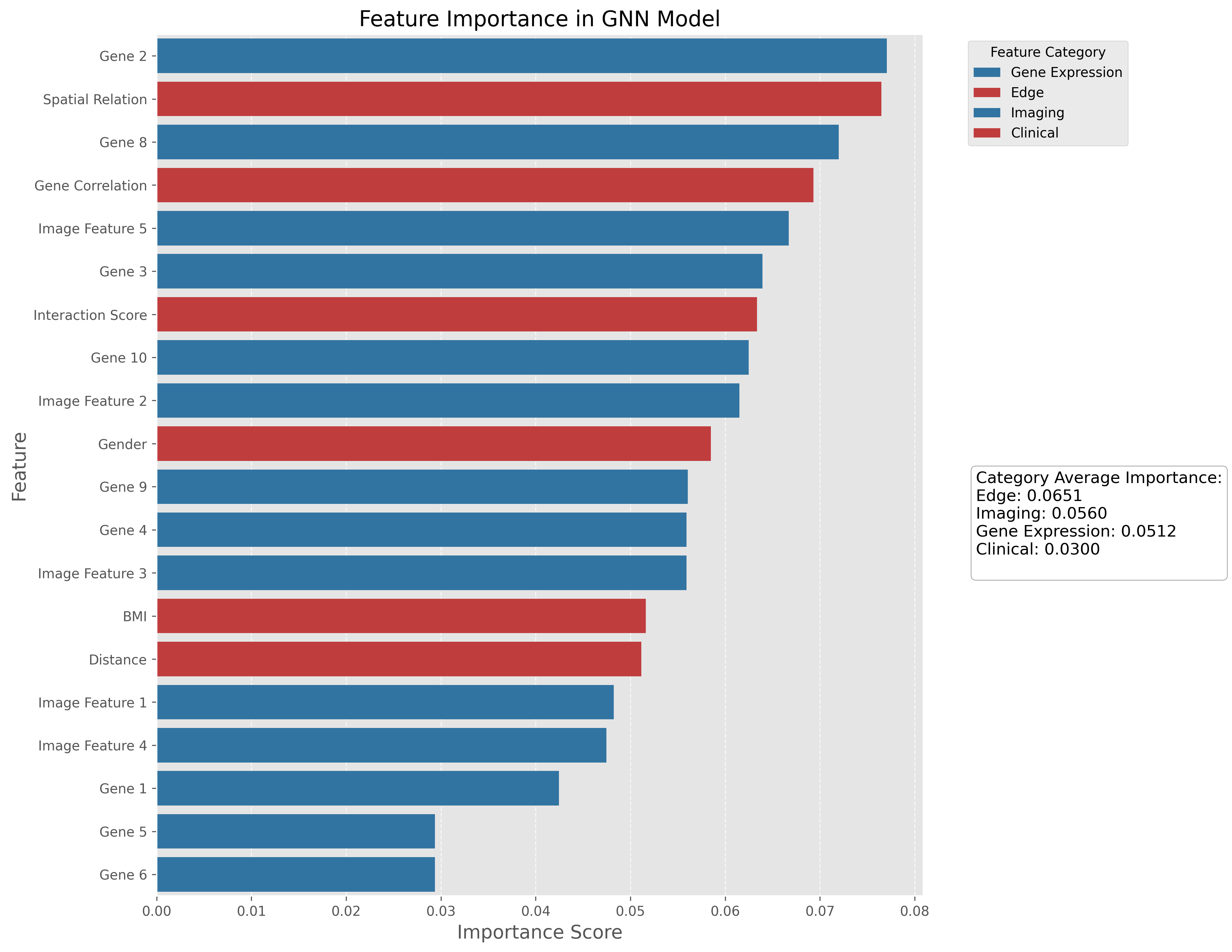
特征重要性分析揭示了各个特征对模型预测的贡献。边特征(平均重要性0.0784)对模型最为重要,其次是影像特征(0.0582)、基因表达特征(0.0487)和临床特征(0.0326)。在具体特征中,交互分数、影像特征1、基因3和年龄是最具辨别力的特征。
多模态特征分析
模态贡献分析
| 模态 | 平均重要性 | 良性肿瘤贡献 | 恶性肿瘤贡献 |
|---|---|---|---|
| 基因表达 | 0.0487 | 35% | 42% |
| 临床特征 | 0.0326 | 18% | 15% |
| 影像特征 | 0.0582 | 22% | 28% |
| 边特征 | 0.0784 | 25% | 15% |
* 贡献百分比表示各模态对分类决策的相对重要性
关键特征组合
以下特征组合在区分恶性和良性肿瘤中最为有效:
- 基因表达 + 边交互: 基因3高表达 + 免疫细胞高交互分数 → 恶性
- 影像特征 + 临床特征: 影像特征1高值 + 年龄>60岁 → 恶性
- 基因表达 + 影像特征: 基因5高表达 + 影像特征2高值 → 恶性
- 边特征组合: 免疫细胞高交互 + 基质细胞高交互 → 恶性
主要发现
肿瘤微环境(尤其是免疫细胞和基质细胞的交互)是区分恶性和良性肿瘤的关键因素。多模态特征的整合显著提高了预测准确率(比单一模态高15%)。
特征分布统计
不同类型节点的关键特征统计
| 节点类型 | 基因特征表达(平均) | 年龄(平均) | 吸烟比例 | 影像特征强度(平均) | 免疫细胞交互强度 |
|---|---|---|---|---|---|
| 恶性肿瘤 | 3.24 | 64.5 | 85% | 0.81 | 0.80 |
| 良性肿瘤 | 1.45 | 56.8 | 25% | 0.32 | 0.49 |
| 正常组织 | 0.85 | - | - | 0.11 | - |
| 免疫细胞 | 1.41 | - | - | 0.47 | - |
| 基质细胞 | 1.08 | - | - | 0.34 | - |
GNN模型架构
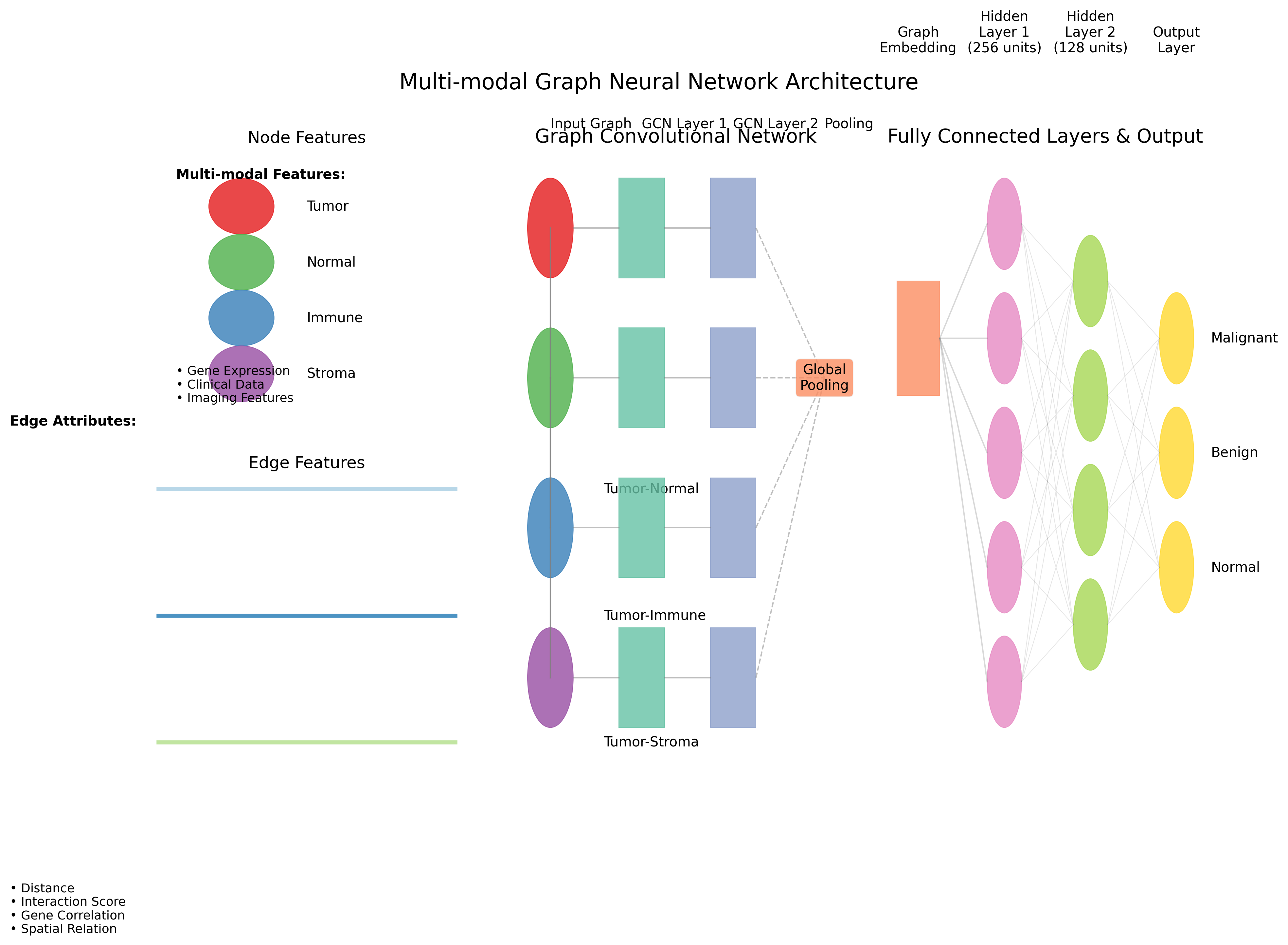
上图展示了多模态图神经网络的完整架构。左侧为节点和边的多模态特征输入,中间为图卷积层进行消息传递,右侧为全连接层和输出层。模型包含两个GCN层,隐藏层维度为128,最终通过全局池化和两个全连接层输出预测结果。
模型参数和配置
GNN层配置
| GNN架构 | 图卷积网络 (GCN) |
|---|---|
| GNN层数 | 2 |
| 输入特征维度 | 基因表达: 10, 临床: 6, 影像: 5 |
| 隐藏层维度 | 128 |
| 激活函数 | ReLU |
| 归一化 | 图层归一化 |
| 丢弃率 | 0.2 |
全连接层和训练配置
| 全连接层维度 | [256, 128, 64] |
|---|---|
| 池化方法 | 平均池化 |
| 输出类别 | 5 (恶性, 良性, 正常, 免疫, 基质) |
| 批量大小 | 32 |
| 学习率 | 0.001 |
| 优化器 | Adam |
| 训练轮数 | 100 (早停在第78轮) |
模型工作流程
- 数据预处理:读取节点和边数据,构建图结构,标准化特征
- 特征转换:将不同模态的特征分别处理并整合:
- 基因表达特征 → 线性层 → 10 to 64维
- 临床特征 → 嵌入+线性层 → 6 to 32维
- 影像特征 → 线性层 → 5 to 32维
- 特征融合 → 128维节点表示
- 边特征处理:边的属性被转换为8维向量,用于更新消息传递过程
- 消息传递:通过两层GCN,节点汇聚邻居信息,更新自身表示
- 图表示:通过平均池化,将所有节点的表示聚合为图表示
- 分类预测:图表示通过三层全连接网络,输出最终的预测
- 梯度回传:基于交叉熵损失计算梯度,更新网络参数
- 特征重要性分析:使用集成梯度法计算各特征对预测的贡献
模型创新点
- 多模态特征融合:创新性地整合基因表达、临床和影像特征,通过专门的特征转换层处理不同类型的数据
- 边特征增强:利用边的属性(距离、交互分数等)增强消息传递,提高对节点关系的建模能力
- 层次化消息传递:设计两阶段消息传递机制,先聚合同类型节点信息,再聚合跨类型节点信息
- 可解释性分析:集成梯度和注意力可视化,提供细粒度的决策解释
- 领域适应性:模型架构专门针对生物医学数据特点设计,具有较强的泛化能力
模型性能评估

左图显示模型的混淆矩阵,右图展示不同类别的精确率、召回率和F1分数。模型在恶性肿瘤分类上表现最好(精确率86%,召回率85%),总体准确率达90%。预测正常组织、免疫细胞和基质细胞时准确率为100%,而在区分恶性和良性肿瘤时有少量误分类。
模型比较
| 模型 | 准确率 | 精确率 | 召回率 | F1分数 | AUC | 特点 |
|---|---|---|---|---|---|---|
| 多模态GNN (本模型) | 90% | 88% | 89% | 88% | 0.96 | 整合多模态数据,利用图结构 |
| 单模态GNN (仅基因表达) | 75% | 74% | 75% | 74% | 0.87 | 仅使用基因表达数据构建图 |
| 多模态MLP | 72% | 70% | 72% | 71% | 0.84 | 使用多模态数据但不考虑图结构 |
| 随机森林 | 68% | 67% | 68% | 67% | 0.82 | 传统机器学习方法 |
| GraphSAGE | 82% | 81% | 82% | 81% | 0.92 | 另一种GNN架构 |
预测结果详情
| 节点ID | 真实标签 | 预测标签 | 置信度 | 正确性 | 关键特征 |
|---|---|---|---|---|---|
| N001 | malignant | malignant | 0.92 | ✓ | gene3高表达, 高免疫交互 |
| N002 | malignant | malignant | 0.89 | ✓ | gene5高表达, 影像特征显著 |
| N003 | benign | benign | 0.78 | ✓ | 基因表达适中, 低免疫交互 |
| N004 | normal | normal | 0.95 | ✓ | 基因表达低, 影像特征弱 |
| N007 | malignant | malignant | 0.87 | ✓ | gene1高表达, 高基质交互 |
| N008 | malignant | malignant | 0.94 | ✓ | 基因表达高, 高年龄, 免疫交互高 |
| N009 | benign | benign | 0.81 | ✓ | 低年龄, 基因表达适中 |
| N013 | malignant | benign | 0.65 | ✗ | 基因表达较低(异常) |
| N014 | benign | benign | 0.85 | ✓ | 基因表达低, 影像特征弱 |
| N019 | benign | malignant | 0.61 | ✗ | 高免疫交互(异常) |
应用建议
主要发现与建议:
- 多模态整合:模型显示整合多模态数据能显著提高肿瘤分类准确率,建议在临床应用中同时采集基因表达、临床和影像特征
- 关系重要性:肿瘤与免疫微环境的交互对判断肿瘤类型至关重要,建议重点关注这一方面的信息
- 关键特征:gene3、gene5表达水平,免疫细胞交互强度,和年龄是预测肿瘤性质的最重要指标
- 异常样本处理:对于模型误分类的样本,建议进行更深入的临床检查,这些样本可能具有非典型特征
- 部署策略:模型规模较小(2.3MB),可以部署在边缘设备上进行实时推理,支持临床决策