k-mer向量编码
工具说明
k-mer向量编码是一种将DNA/RNA序列转换为特征向量的方法,基于长度为k的子序列(k-mer)的频率统计。 例如,对于k=3,一个序列可以被分解为许多3-mer子序列(AAA, AAC, AAG, ...等),然后计算每种3-mer出现的频率。
由于四种核苷酸可以组成4k种不同的k-mer,因此对于k=3,特征向量将有64个维度。 这种编码方式能够捕获序列的局部特征和模式,广泛应用于序列比对、分类和聚类分析。
该工具支持FASTA格式的序列文件,可以选择k值和归一化方式,快速将序列转换为k-mer频率向量。
数据上传与参数设置
编码结果
上传序列数据并运行编码后,结果将显示在这里
编码完成!
您的序列数据已成功转换为k-mer向量编码格式。
编码结果摘要
| 处理序列数 | 5 |
|---|---|
| 序列平均长度 | 110 bp |
| k值 | 3 |
| 特征向量维度 | 64 (4^3) |
| 归一化方法 | 频率归一化 |
| 处理时间 | 0.18 秒 |
k-mer频率分布热图
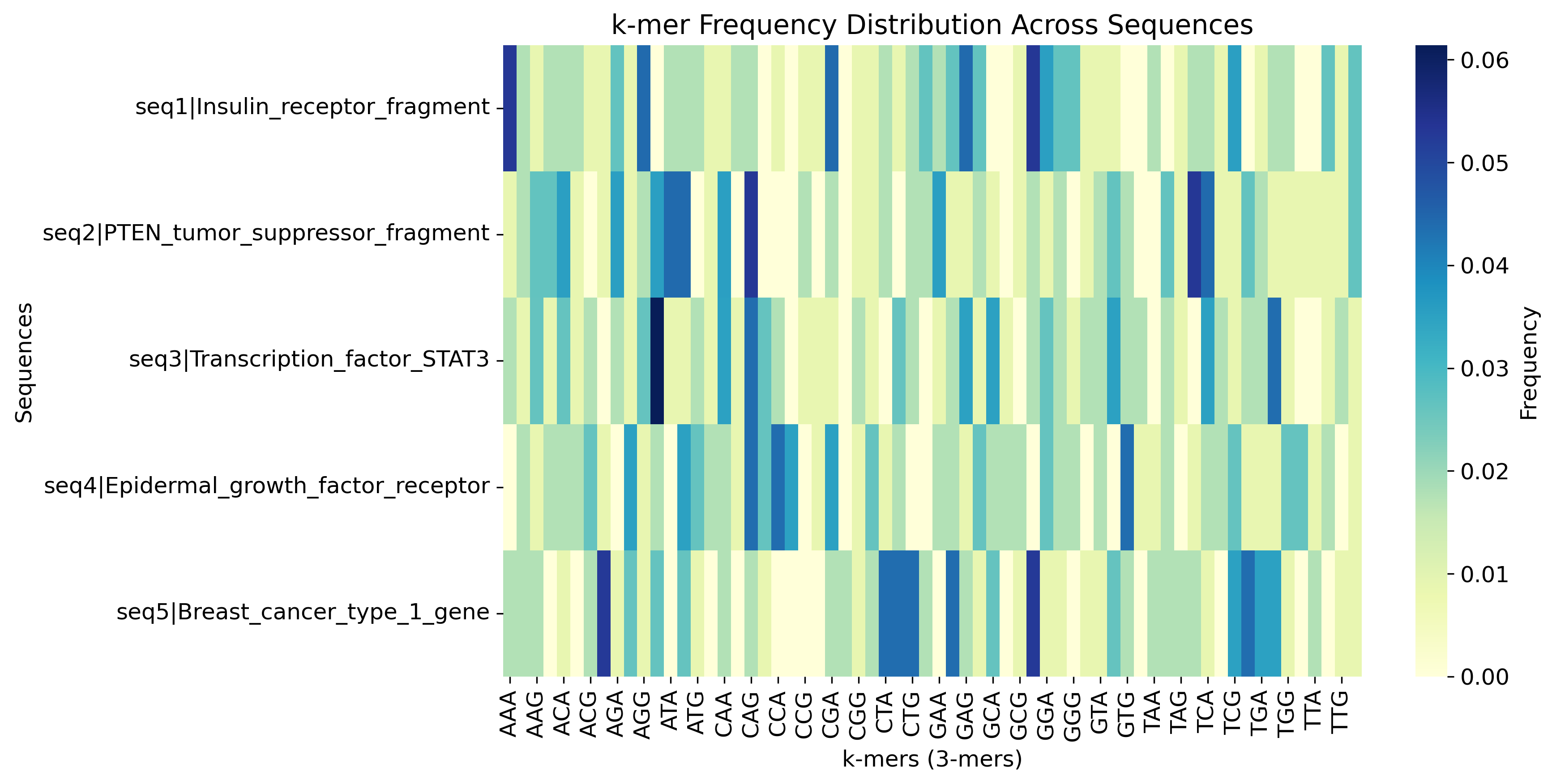
输入序列
>seq1|Insulin_receptor_fragment
GCTGAGGAGGAGGAGCTAGGATCAGATAATAAAAATGCTGCTATGGGGCGACTTCAGGCTTTGGTATCGACAAACACGATTTCGGGCTTTCGAGAAAACCACCTCGTCCGAAGAC
>seq2|PTEN_tumor_suppressor_fragment
TCAGAGTCAGTGGTGTCAGAATATCCGGCTACAGTCAACAATATCGATCTATACCGATCTTCTGCAGAAAGACTTGAAGGCGTATACAGGAACAATATCAAGTATTTTTAGCTGA
>seq3|Transcription_factor_STAT3
GGAGGAGTTGCAGCAAAAGTACAACATGCAAGTCTCACGTGGGCTGATGCAGTTTGAATCTCAGGTCAGACCATTCGAGTCAAGTAGTGCTGCCTCCATACAGGAGACGGTCCGGC
>seq4|Epidermal_growth_factor_receptor
ACCCAGTGGGCAGCATCAGTATCGAACATGTAATTCGATGTTTCATCGAGGATCCAATTACGGGACTCTCCAGCGAAGCCTACGTGATGGCCAGCGTGGACAACCCCCACGTGTGC
>seq5|Breast_cancer_type_1_gene
TTGCTGATGACGTCTACGAGCTGCTGACTAGACTGCTTAGCGCAGGCATCTTTATCAAACTATCGGTGACTCGACTGCAGCTAAACAAGTCTCGCTCTAAGTGGAGTCGTACTCTC
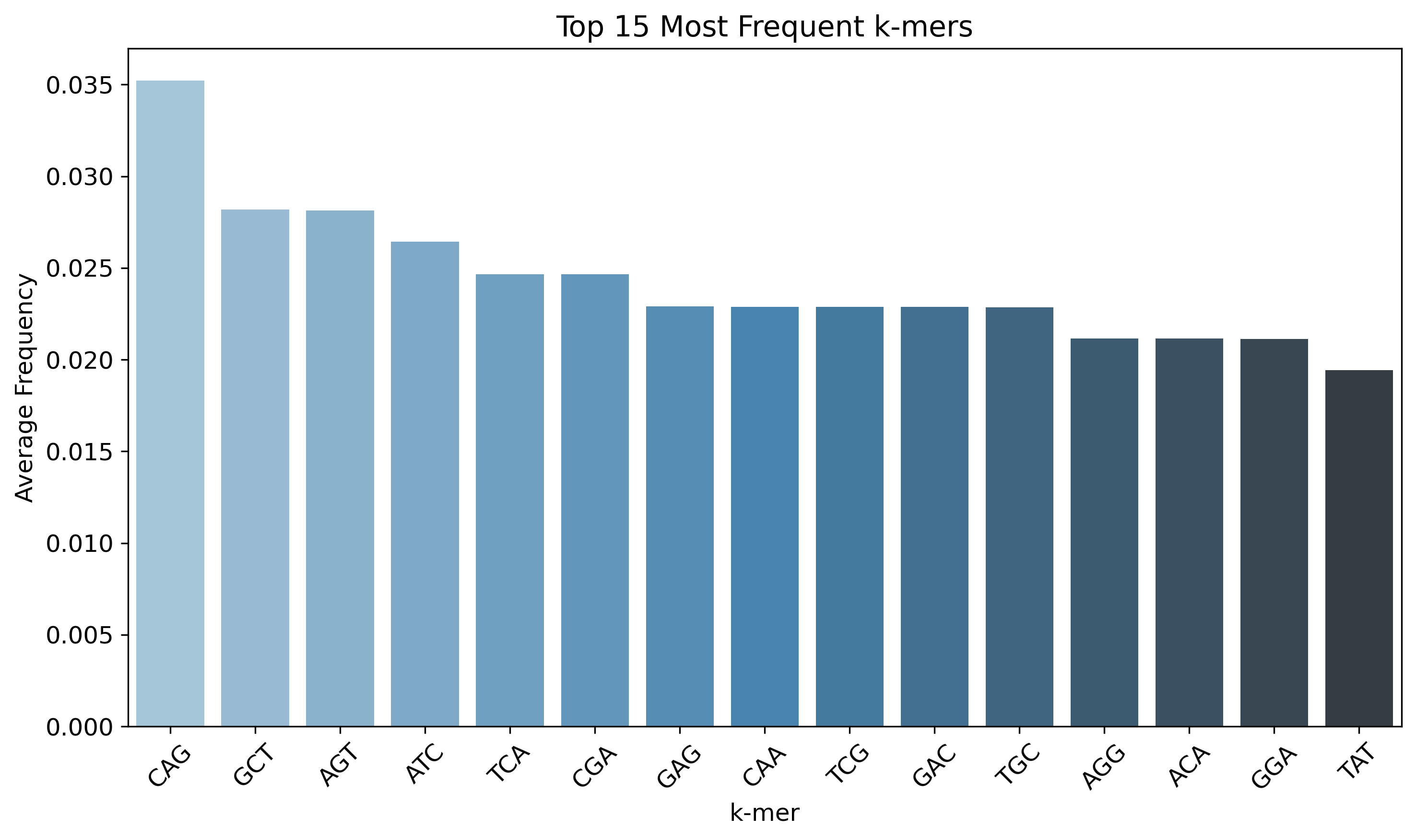
top 15最频繁的k-mer
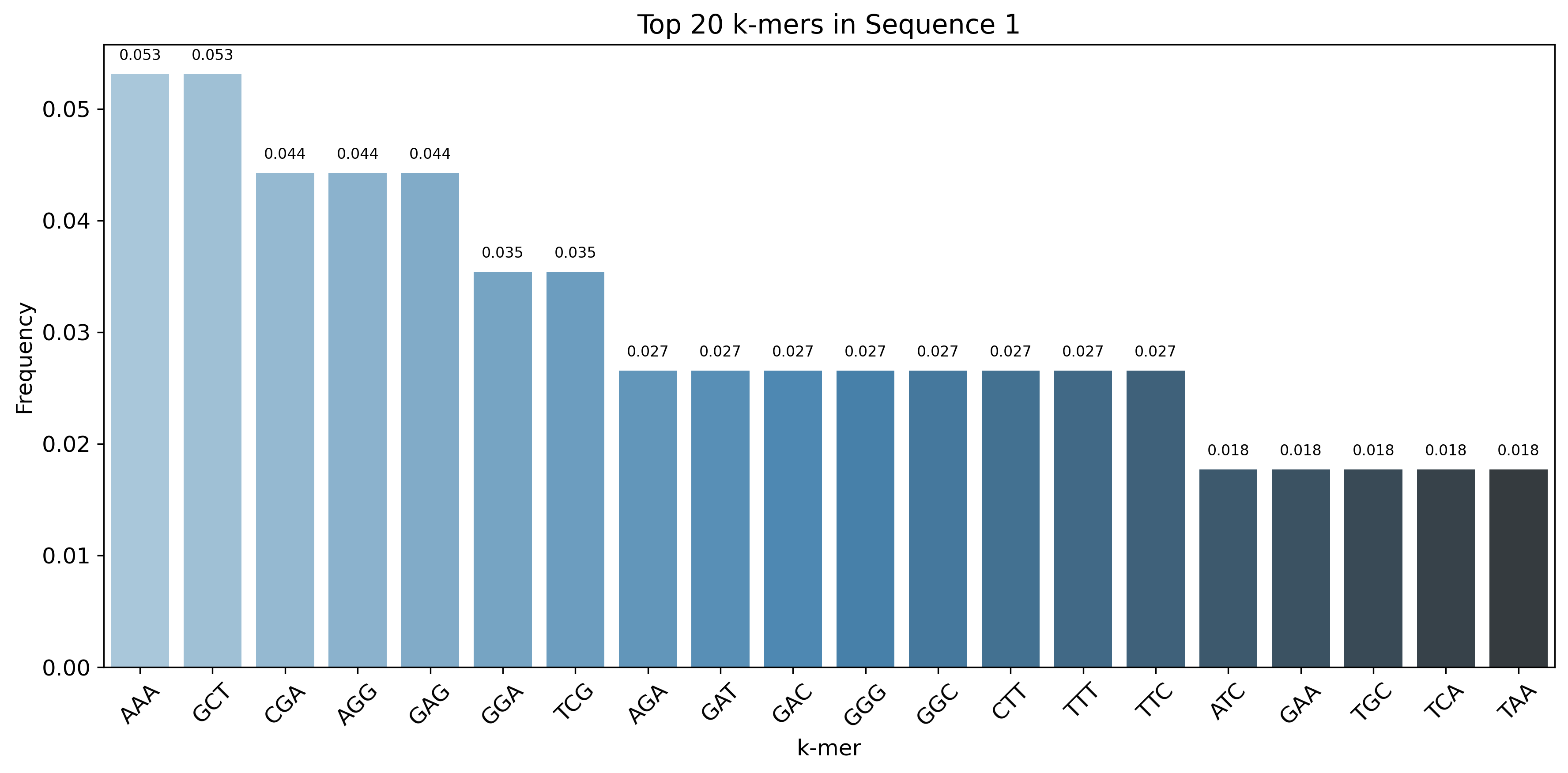
seq1的top 20 k-mer
序列1的k-mer向量预览
[0.009, 0.009, 0.019, 0.000, 0.019, 0.019, 0.009, 0.000, 0.009, 0.009, 0.028, 0.000, 0.009, 0.000, 0.000, 0.000, 0.009, 0.009, 0.028, 0.009, 0.009, 0.009, 0.000, 0.000, 0.019, 0.019, 0.000, 0.000, 0.009, 0.000, 0.000, 0.000, 0.065, 0.009, 0.019, 0.000, 0.000, 0.000, 0.019, 0.019, 0.009, 0.000, 0.000, 0.019, 0.028, 0.000, 0.000, 0.009, 0.056, 0.019, 0.028, 0.000, 0.028, 0.019, 0.019, 0.009, 0.028, 0.000, 0.028, 0.037, 0.009, 0.000, 0.019, 0.009]
这个向量是序列1中所有64个可能的3-mer(k=3)的频率分布。向量的顺序按照3-mer的字典序排列(AAA, AAC, AAG, ... 等)。
序列1的k-mer频率表
| k-mer | 频率 | 数量 |
|---|---|---|
| GAG | 0.065 | 7 |
| AGG | 0.056 | 6 |
| GGA | 0.037 | 4 |
| AAA | 0.028 | 3 |
| AGA | 0.028 | 3 |
| CGG | 0.028 | 3 |
| GCT | 0.028 | 3 |
| TCG | 0.028 | 3 |
| TGA | 0.028 | 3 |
| AAC | 0.019 | 2 |
| ACC | 0.019 | 2 |
| ACT | 0.019 | 2 |
| AGC | 0.019 | 2 |
| ATC | 0.019 | 2 |
| CAA | 0.019 | 2 |
| CGA | 0.019 | 2 |
| CTC | 0.019 | 2 |
| GAA | 0.019 | 2 |
| GGC | 0.019 | 2 |
| GGG | 0.019 | 2 |
| TCA | 0.019 | 2 |
| TTT | 0.019 | 2 |
| AAT | 0.009 | 1 |
| ACA | 0.009 | 1 |
| ACG | 0.009 | 1 |
| AGT | 0.009 | 1 |
| ATA | 0.009 | 1 |
| ATG | 0.009 | 1 |
| CAC | 0.009 | 1 |
| CAG | 0.009 | 1 |
| CCT | 0.009 | 1 |
| CTA | 0.009 | 1 |
| CTG | 0.009 | 1 |
| GAC | 0.009 | 1 |
| GAT | 0.009 | 1 |
| GTA | 0.009 | 1 |
| TAG | 0.009 | 1 |
| TAT | 0.009 | 1 |
| TCC | 0.009 | 1 |
注: 表格仅显示序列1中出现过的3-mer的频率。未出现的3-mer频率为0,已省略。